Método para medir la resistencia bacteriana en infecciones de vías urinarias
Científicos del California Institute of Technology mejor conocido como Caltech han desarrollado un método de prueba que permite identificar a los médicos si la bacteria que se hospeda en sus pacientes es resistente a los antibióticos.
Aunque ya existen análisis clínicos especializados para conocer este dato, obtener los resultados puede llegar a tardar hasta tres días en comparación con este nuevo instrumento que ofrece resultados confiables en menos de 30 minutos.
La resistencia a los antimicrobianos no solo se genera en bacterias sino también en virus, hongos y parásitos. Es un proceso natural generado normalmente a partir de mutaciones genéticas en los microorganismos ocasionados por la exposición prolongada a antibióticos, antifúngicos o antivirales según sea el caso. Sin embargo el uso incorrecto y abuso de los antimicrobianos ha acelerado el proceso de mutación.
Este tema se ha convertido en un problema de salud pública mundial que de no ser estudiado y controlado podría llegar a generar complicaciones en intervenciones médicas como las cirugías y ocasionar consecuencias negativas en la economía de los pacientes al tener que invertir mayor cantidad de dinero en fármacos durante mayores periodos de tiempo. Incluso se sabe existen ya cepas bacterianas que presentan resistencia múltiple a varios antibióticos
Como consecuencia de esto en ocasiones los tratamientos médicos son recetados y aplicados en el paciente sin conocer la tasa de éxito de acuerdo al ente infeccioso. La respuesta a esto podría estar en realizar análisis para conocer la resistencia del microorganismo, sin embargo no es útil en casos de urgencia cuando se debe esperar por días para conocer los resultados clínicos.
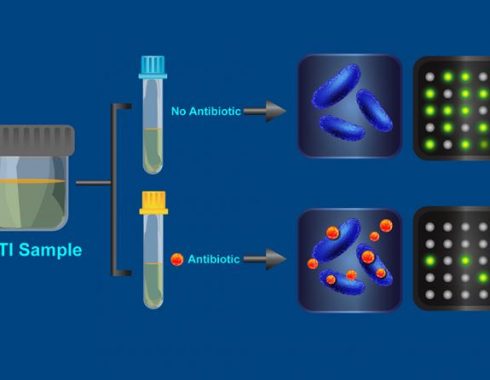
Para solucionar dicho problema los científicos de Caltech diseñaron su innovadora prueba que permite obtener esta información en minutos con una tasa de confiabilidad del 95% comparada con pruebas estándar de resistencia bacteriana. En la fase de experimentación y comprobación de su investigación enfocaron su método en el análisis de infecciones de vías urinarias a través de la resistencia de la bacteria Escherichia coli haciendo uso de 54 muestras de orina.
El proceso por el cual realizaron esto fue dividir en dos partes cada prueba individual de orina infectada con Escherichia coli. Un parte fue expuesta a un antibiótico durante 15 minutos mientras la otra parte permanecía sin alteraciones.
Luego de esto las bacterias en cada muestra fueron analizadas a través de un proceso de detección química llamado amplificación isotérmica mediada por loop en tiempo real que permite amplificar marcadores específicos de ADN. Esto permite observar y contabilizadas dichos marcadores a través de un chip y que los representa como puntos fluorescentes en una pantalla.
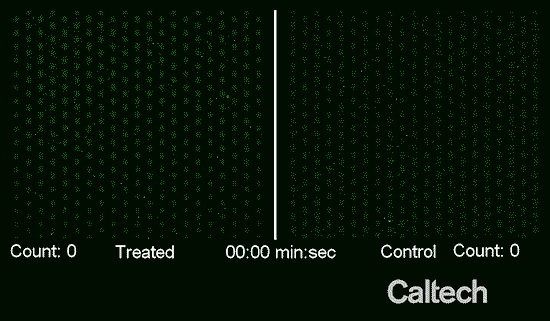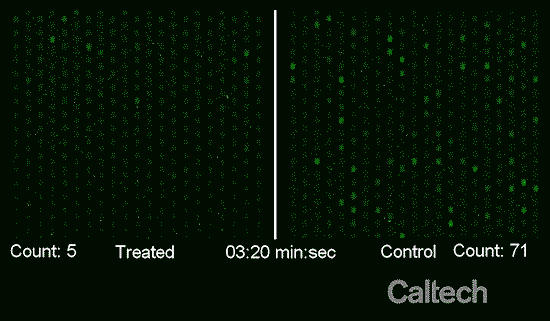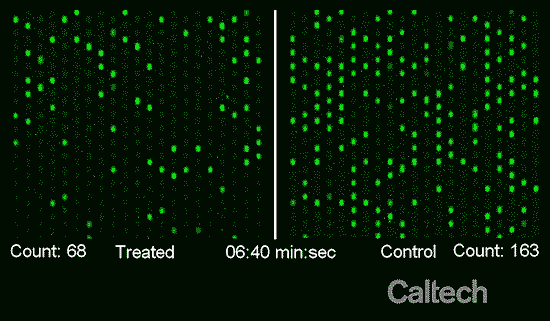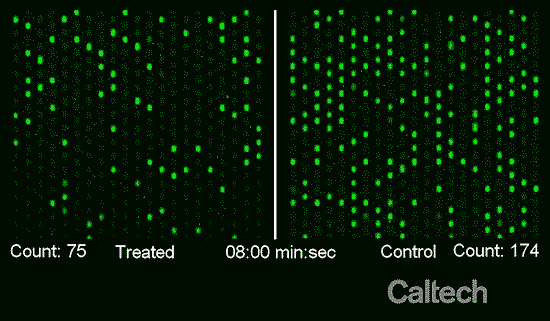
Fuente: Caltech
De acuerdo a los resultados sobre el número de puntos fluorescentes correspondientes a cada parte de las muestras se puede saber si la bacteria es resistente al antibiótico aplicado. En el caso de la muestra tratada se esperaría obtener un número menor de puntos que la mitad de muestra no tratada. Sin embargo en el caso de no existir diferenciación entre ambos resultados indicaría una resistencia bacteriana por parte del microorganismo hacia el antibiótico.
Atender este problema es vital para la sociedad actual pues para 2050 podría llegar a generar 10 millones de muertes y un impacto económico de un trillón de dólares. Países como México consideran la resistencia microbiana como un problema al nivel de enfermedades como la tuberculosis, sida o cáncer.
Siendo de tal magnitud el problema los científicos de Caltech pretenden colaborar a mejorar la situación mundial a partir sus investigaciones y de un próximo desarrollo que sería compatible para distintos patógenos, muestras y antibióticos para acelerar la toma de decisiones médicas, así como mejorar el manejo control de enfermedades infeccionas y resistentes.
La investigación que respalda dicho desarrollo se titula Rapid pathogen-specific phenotypic antibiotic susceptibility testing using digital LAMP quantification in clinical samples y fue presentada en la publicación Science Translational Medicine en octubre de 2017.